Déformation d’images volumiques
Stage M1 ou M2 pro, encadré par François Faure
Contexte
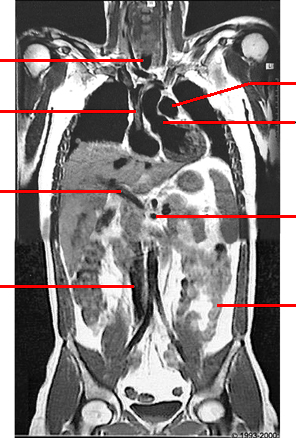
L’imagerie médicale permet de visualiser l’intérieur du corps pour comprendre des pathologies ou préparer des interventions. Pour montrer les muscles et organes déformables, elle se pratique sur des patients allongés. Dans cette position, la colonne vertébrale s’applatit et prend une forme différente de la position debout. Dans certains cas on aimerait avoir des images du corps en position debout, mais les machines d’imagerie qui le permettent modélisent seulement les os, pas les organes mous. Il faudrait donc pouvoir déformer l’image allongée pour la faire correspondre à la forme de colonne vertébrale debout.
 |
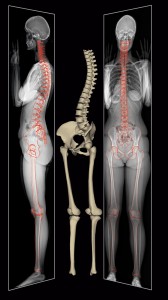 |
| Image IRM, en position couchée. | Images et reconstruction EOS, position debout. |
Notre méthode de Transfert Anatomique, illustrée ci-dessous, calcule une déformation qui pourrait s’appliquer à l’image allongée, pour la faire correspondre à la forme debout. Il ne manque plus qu’un module d’affichage d’image volumique déformée.
Travail demandé
Le but de ce stage est d’implémenter une méthode d’affichage d’image volumique déformée. Elle devra être suffisamment rapide pour traiter plusieurs centaines de millions de voxels par seconde. On pourra s’inspirer d‘une méthode proposée récemment sur GPU.
Profil
- Niveau M1 ou M2 d’informatique ou mathématiques appliquées
- Connaissances ou forte motivation pour la programmation GPU
Contact: François Faure

